
Macot参数设置
通过网页端(http://www.matrixscience.com/search_form_select.html)访问Mascot,可以看到三种不同的搜库模式,如下所示:

上图中图左为Peptide Mass Fingerprint(PMF),中间的图为Sequence Query,右图为MS/MS Ion Search。
Peptide Mass Fingerprint(PMF):PMF检索主要应用于只有一系列MS1信息的蛋白鉴定,主要应用于MALDI数据分析。
Sequence Query:可以对肽段的质谱数据进行序列和可能组成分子量的混合搜库,来自MS/MS,难以完整检索序列,是一种序列标签查询超级集合。
MS/MS Ion Search:使用未经注释的大量MS/MS图谱,进行数据库肽段匹配,但原始谱图必须转化为谱峰列表。
三种搜库方法界面大多数参数是共通的,其中sequence query的界面和另外两个搜库界面的主要区别是,它没有数据上传的选项,而需要输入文本形式的查询信息。搜库界面的每一项参数的名称都包含帮助文档的链接,点击后可以直接查看具体的说明。
具体参数说明:
1、用户信息:

Mascot支持多用户管理和登录,以及自动发送结果完成信息到用户邮箱,因此填写这些信息能够使您的搜库结果更容易查找和管理。
2、数据库
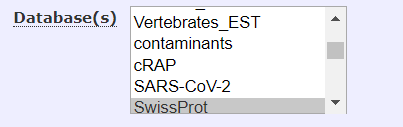
Mascot公共服务器上只有有限的几个常用数据库,本地运行时您可以任意添加数据库,一个数据库可能包含单一物种的序列,也可能包含大量物种信息,您可以使用Mascot的Taxnomy filter功能来选择数据库中特定物种的序列进行搜库。当您还不清楚该物种是否包含比较完整的物种序列信息时,千万不要选择taxonomy filter进行搜库。
当您无法获知数据库中样本的准确信息时,使用Swiss-Prot是一个合适的起点。该数据库中的序列经过人工审核,质量较高且基本都是注释信息。当您的目标生物的蛋白序列已经被很好的注释,如人、小鼠、酵母或拟南芥等,您就没必要选择Swiss-Prot以外的数据库了。如果是其他物种,您也可以首先访问Uniprot.org来确定Swiss-Prot数据库中是否包含其完整的蛋白序列信息。
当您研究的是非模式生物,且为细菌或植物时,Swiss-Prot往往信息量较少,这时您需要寻找一个序列更完整的数据库进行搜库。最著名的两大完整的蛋白序列数据库是NCBInr和UniRef100.如果您的研究对象连基因组都未测序,那么您可以尝试搜索一系列表达序列标签(EST)数据库来评估搜库可能性。
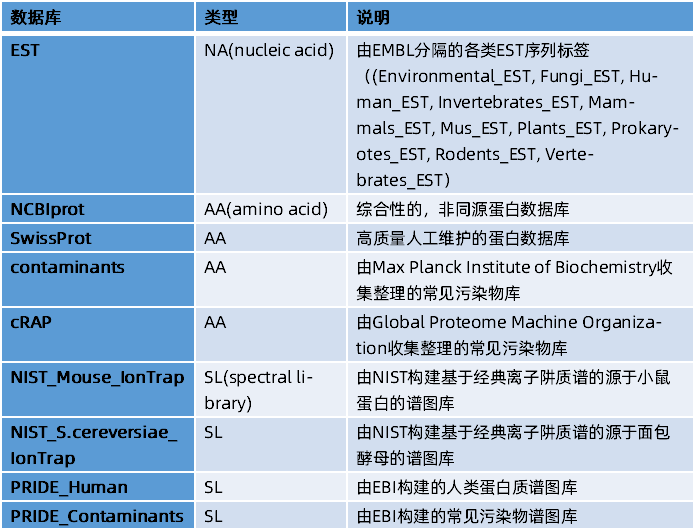
Mascot公共服务器上数据库总结如下:
Note:
•对于肽质量指纹谱,谱图库和NA数据库都是不可用的。用EST序列来对多肽质量数搜库是无意义的,因为这些条目都是短序列而不是完整蛋白。
•对于MS/MS search, 在公共的Mascot server上你应该先用蛋白数据库做一遍搜库,再尝试用EST数据库搜库。
•多库联合搜索,添加污染物库相当有意义。
•在Mascot2.6的新版本中,新添加的Spectral library搜索功能是基于NIST的MSPepSearch搜索引擎进行的,spectral library和序列数据库搜库结合的更好,提供了更严格的蛋白推断结果。
如果蛋白数据库搜索没有产生阳性匹配的结果,结果页面上会提示你用EST数据库再次搜库。同时加入contaminants数据库搜索,可以更好地帮助您判断实验结果的好坏,也可以更容易地发现人细胞样本被BSA所污染的情况。就算您做的是大规模的全蛋白质组分析,您也会关心样本是否被Serum Albumin、Keratin等干扰。在Mascot搜索时,使用control按键,可以轻松地选择2个甚至于更多数据库同时搜索。如果您搜索的是多物种数据库并使用taxonomy filter自动进行物种选择时,你也不用担心造成混乱,因为该选项只会对设置了多物种索引的多物种数据库进行筛选,而contaminants数据库等会自动跳过筛选。过去对于database 的类型是有限制的,但是在2.6的新版本中,这个限制已经被取消。
3、Taxonomy 物种信息
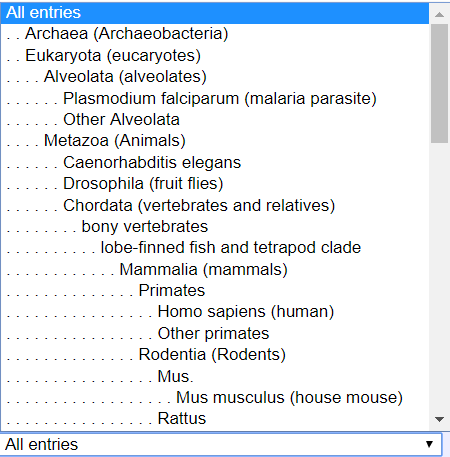
我们可以使用大数据库进行搜库,在需要使用特定类型的物种时,在Taxonomy中进行过滤即可,另外如果发现下拉菜单中的物种分类列表不包含您所需要分析的物种时,可以使用文本编辑器打开Mascot安装目录下config子目录中的Taxonomy文件,将从NCBI或者Uniprot中找到的物种分类Taxonomy ID加入其中即可。使用NCBInr或者Uniref100时请尽量保证互联网畅通,这样在这两个数据库有新序列更新时,mascot可以很容易的同步更新新蛋白的物种索引信息。
Note:注意选择的物种分类是否包含足够数量的候选序列,若选择的目标物种序列数量太少,则需要考虑选择相似物种进行搜索。另外如果该界面中没有您想找的物种,可以去数据库官方网站搜索该物种及蛋白序列的数量来评估。
4、Enzyme酶切类型选择

在enzyme下拉菜单中包含大多数常用酶,因此您只需选择实验中使用的对应酶切类型即可。当选择实验用的酶时,请注意低特异性的酶可能会消化出大量短肽,这样的样品进行质谱分析可能会造成大量非特异性匹配。肽段越长,对于鉴定的效率越高。大多数情况下,Trypsin酶切产生的序列最适宜进行质谱分析和搜库,通常1200Da到4000Da之间的肽段鉴定效率最高。
NOTE:一般情况下Trypsin极少发生漏切,因此漏切数目一般选择1个。如果有大量的漏切结果,可尝试将漏切设置提高到2,特殊情况下也尽可能选择半酶切而不要选择非酶切。非特异性酶切只适用于多肽组学研究,且不能用于PMF搜索。

上面这张表格是对同一个数据集进行特异性酶切、半酶切和非酶切搜库的结果,非酶切的搜库耗时远远高于正常酶切和半酶切,而匹配到的正确结果数量反而下降了。因为非酶切搜库带来大量随机匹配结果,并且可能比正常酶切肽段得分更高。因此,在Mascot中用非酶切搜库并不是一个好主意,除非您的样本的确是这个样子的,某些需要进行非酶切搜库的情况下,半酶切更适合进行质量评估。
当研究的是内源性多肽时,只能选择Enzyme为none搜索所有可能的酶切方式。注意NoCleave是完全不进行酶切分析和None完全相反,用于Top Down分析。
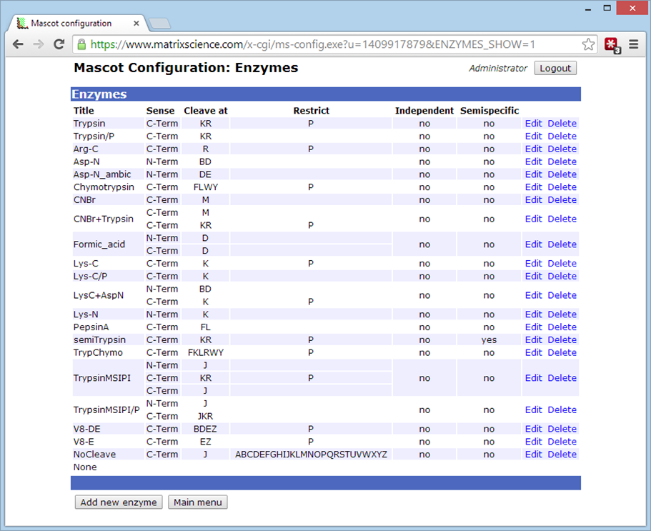
上图为Mascot中酶切参数设置界面。所有信息都可以进行编辑或者新建酶切类型,设置内容包括酶切位点,酶切末端,限制性位点等,Mascot支持两类混合酶切定义。Independent类型的混合酶切用于多种酶分别进行酶切后混合检测的样本,比如同一个样本分成两份,一份trypsin酶切,一份Asp-N酶切后混合分析。而非Independent类型指得是同一个样本先后或同时进行混合酶切。比如Trypsin联用CNBr酶切。
5、Modification修饰类型设置
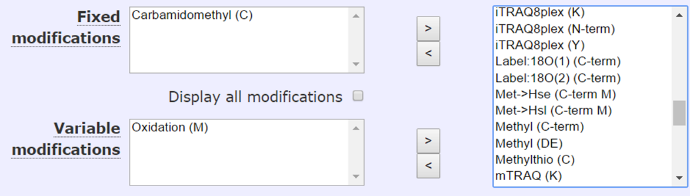
上图是Mascot修饰设置界面,修饰分为两种类型,固定修饰和可变修饰。
第一种为fixed或用于标记定量的修饰,指的是所有该类氨基酸上一定会发生的修饰。如还原烷基化cysteine的carbamidomethyl修饰。或者iTRAQ,SILAC的末端标记修饰等。通常此类修饰不会对搜库结果造成明显干扰,也不会降低搜索速度。
第二种是可变修饰,是指可能会发生,也可能不会发生在该类型氨基酸上的修饰,只有非常丰度的可变修饰搜库才有意义。如磷酸化、酰化等修饰。
同一个残基不能设置两种类型修饰。如果在同一个残基上设置了一个固定修饰和一个可变修饰,将不会搜到非修饰结果。蛋白质的翻译后修饰信息可以在http://ww.unimod.org上找到,同时本地Mascot服务器上您可以任意自定义新型修饰。
固定翻译后修饰搜索不会对结果的灵敏度造成任何影响,因为这只是有和无的差别,而可变修饰类型的选择会对结果的灵敏度有很大影响,我们建议选择样本中高丰度的修饰进行搜库,比如没有富集过磷酸化肽的样本并不值得加入磷酸化可变修饰搜库。
Mascot 限制一次搜库中可变修饰的数目:最多9个variety modification
为何不能同时搜索所有潜在修饰?
●一共有400多种可能的潜在修饰,而大多数修饰含量都非常低
●搜库时加入这些修饰会大大增加搜库时间,并导致结果假阳性率大大升高
●一般采用多轮搜库策略来进行可能的修饰寻找
6、Quantitation 定量方法

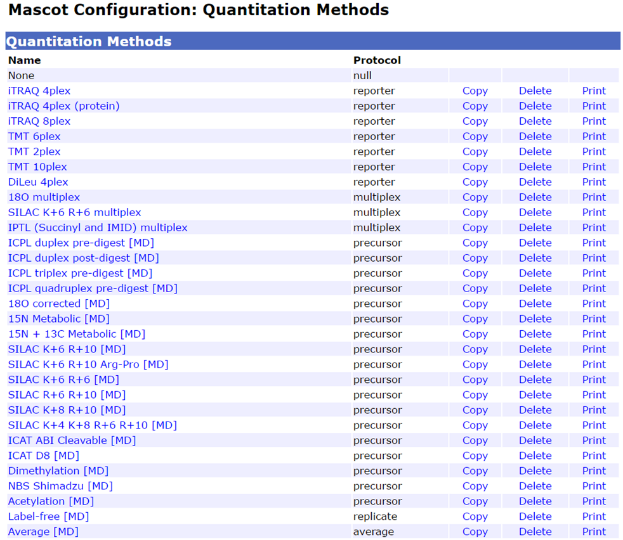
在进行搜库参数设定时,Mascot将常用的定量方法及其修饰情况集成到了Quantitation选项中,下拉菜单中选择对应的定量方法,无须在修饰类型中设定定量相关的修饰。上图为Mascot提供的定量方法,其中所有定量方法后带有[MD]字样的方法均需要联用Distiller进行处理,网页中直接提交将无法定量。点击相应的定量方法可以进行编辑,或者点击右侧的Copy可以当前方法作为模板进行新方法的设计。
7、Mass tolerance质量容差
在Mascot搜库界面中。PMF与Seuquence和MS/MS Ions search两种搜库方法的设置不同。在PMF界面中为以下设置项:

包含Protein mass和Peptide tolerance两项设置。
Protein mass该选项主要用于Top-down研究,设置蛋白质量数搜索的上限。如果留空的话,则使用Mascot可以搜索到的最大质量数进行匹配。这个选项并不常用,但是在某些特殊的研究中有一定的需求,如果分析对象来自于一些序列很长的蛋白碎片,那么它可以一定程度上提高匹配的灵敏度,该选项在Bottom-up的MS/MS搜索中不起作用。
Peptide tolerance:通常根据一起类型和质量精度设置肽段母离子的搜索容差。设置的Tol数值应该大于仪器的实际表现。Tol的容差设置单位:百分比,milli-mass units, parts per million或者Daltons。目前高精度质谱而言,一级最常用的是ppm。
在Seuquence和MS/MS Ions search两种搜库方法的界面中,需要设置肽段母离子和MS/MS二级离子容差。


大部分常用类型仪器的参数设置见上图。
Peptide tolerance设置同PMF中的相应设置。
MS/MS tolerance该选项用于设置MS/MS碎片的质量范围。单位是milli-mass units,Daltons或ppm。该选项设置过高或过低会影响结果离子得分。
#13C 同位素峰校正:有时候质谱原始数据产生的时候,仪器会错误的选择肽段的13C同位素峰作为12C的质荷比,从而会造成母离子质量数1Da的偏离。甚至挑选了13C2 peak,造成2Da的误差。#13C选项可以搜索此类情况的谱图。通常情况下,由于Deamiation修饰非常接近于1Da,不建议该选项和Deamidation同时搜索。
该参数设置为1或2时,Mascot会自动去寻找可能错误分配的13C和13C2的谱峰。计算过程如下:
首先对母离子进行匹配
TOL > absolute(exp - calc) Da
如果该参数设为1,Mascot会进一步搜索
TOL > absolute(exp - calc - 1) Da 范围内可能的母离子匹配
如果设为2,那么会继续搜索
TOL > absolute(exp - calc - 2) Da 内的母离子。
8、Mass Type

Mass Typy指定实验得到的谱峰信息是以average 还是 monoisotopic形式保存的。下图展示了Average和Monoisotopic之间的区别。Monoisotopic 代表了最高丰度的天然同位素峰的准确质量数。Average mass 是化学质量数,代表同位素分布的中心值。错误设置会导致结果误差较大。
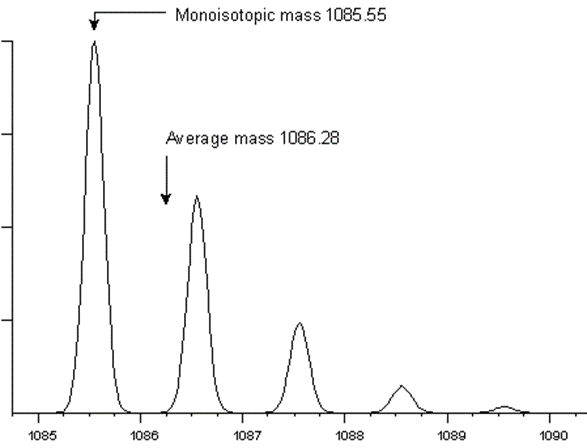
目前大多数高精度质谱都能够给出准确的monoisotopic质量数。除非仪器生产谱图时就已经进行了centroid处理,这样会导致分辨率识别有问题。因此建议仪器设置时MS1的保存格式为Monoisotopic,MS2如果本身精度不高,Average和Monoisotopic影响不大,如LTQ。如果该选项设置错误的话,对于肽段和蛋白,在平均分子量与单同位素分子量之间的差接近0.06%。关于质量精度与分辨率的详细内容可以参考网页http://www.matrixscience.com/help/mass_accuracy_help.html。
9、Charge肽段带电荷情况
Charge选项用于指定肽段的价态。价态被用于根据检测到的质荷比计算母离子的相对分子质量(Mr)。左上图的单选框主要用于PMF搜索,指定保存离子的价态格式。


上图中左图单选框主要用于PMF搜索,指定保存离子的价态格式。右图中的下拉菜单用于MS/MS和Sequence query搜索。下拉菜单中的1+代表的是MH+,而1-代表的是M-H-,2+代表MH2++等。
对于MALDI-PSD的数据,母离子只会是MH+也就是必须指定为1+。对于电喷雾源的数据,一般带多电荷,对于MS/MS搜索,该值一般指定为Default,只有peak list中不包含电荷信息时才需要进行修改。目前大多数质谱仪器的谱图中都会包含母离子的电荷信息,因此该选项基本不用设置。
搜库最佳电荷是2+~3+离子;1+的谱峰过多会导致MS2谱图信息大幅减少;4+以上谱峰过多会导致MS2谱图过于复杂难以解析。
10、Data
上图为PMF的数据上传方式,可以有两种方式数据上传形式。可以直接上传谱峰列表list文件,或者在下方直接输入谱峰信息。PMF的谱峰列表文件比较简单,一般来说就是每行一个质量数,或者额外有强度信息。
Mascot支持的其他格式包括:
Mass [ intensity] [additional text]
Applied Biosystems Data Explorer (.pkm)
Bruker Analysis AutoXecute Data Report
Bruker XML
mzData (1.05)
mzML


上图为MS/MS Ion Search的数据上传界面,必须符合ASCII格式的文件,该格式较为复杂无法自动识别,需要手动指定类型。目前最常用的格式是Mascot generic file MGF。详细的数据格式组成说明参考Mascot在线文档:
http://www.matrixscience.com/help/data_file_help.html
11、Instrument

MS/MS分析中,仪器类型不同得到的谱峰列表会有较大差别。该选项中不同类型的仪器所包含的碎片离子类型不同,对于肽段匹配和结果打分计算均有影响。
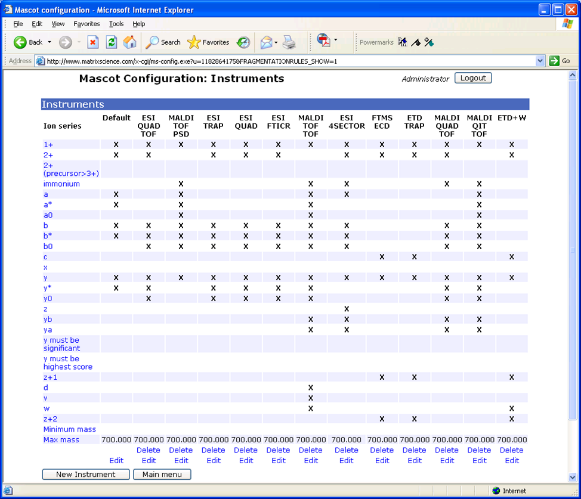
上图为mascot的Configuration-Instruments界面截图,其中“Default”是在旧版本中使用的碎裂类型。根据使用的仪器类型选择对应的碎片模式,同时您也可以修改其中的设置。比如上图中最右列的ETD+W就是修改后增加W离子的ETD个性化设置。设置和您使用的仪器能够产生何种类型碎片离子相关,选择最接近的仪器类型用于搜库即可。
12、Error Tolerant Search

如果您的MS/MS数据可能存在有意义的未知翻译后修饰或者SNP,那么您可以基于当前搜索结果选择非显著匹配的谱图进行进一步的error tolerant 搜索,点击结果界面中的Re-search 然后选择error tolerant,这样的方法可以基于当前正常匹配的蛋白寻找其中可能的未知修饰和SNP。
是寻找可能翻译后修饰、SNP、非特异性酶切的工具。该选项一般用于普通搜库后的第二轮搜库。
第一轮搜库一般选择尽可能少的可变修饰种类,然后留给Error Tolerant来评估可能发生的后修饰类型等。在Error Tolerant搜索中,并不会大大增加搜索时间,因为它使用的search space是基于第一轮搜库的结果,因此该方法可以用于提高序列覆盖度,但不会找到在第一轮搜库中未匹配到的蛋白。因此如果该可变修饰的丰度很高,那么必须加入第一轮搜库才不会导致结果大幅减少。当目标蛋白修饰种类非常多或者每个蛋白可能只有一条能够匹配的蛋白时,error tolerant搜索将无能为力。这主要发生在组蛋白翻译后修饰研究及内源性多肽分析。如果您的目标是分析某个蛋白,那么您需要尽可能多的获取它的肽段谱峰信息,因为导致某张谱图无法成功鉴定的因素很多,所以候选谱图很少的话会降低其检出概率。
多轮搜库策略:
第一轮:尽可能减少可变修饰搜库
第二轮:基于第一轮结果进行Error Tolerant搜库
第三轮:选出高频度潜在修饰进行搜库,突变数据单独提出分析
13、Decoy

在进行MS/MS数据搜库参数设置时选中Decoy后,可以对结果进行false discovery rate错误发现概率评估。对于大规模蛋白质组学数据分析来说,评估结果的False Discovery Rate非常重要。目前较主流的评估手段为:Target-Decoy搜库方法。Target-Decoy搜库的结果结合Percolator机器学习打分重新估算FDR值可以有效改进大规模数据集的灵敏度。
缺陷:无论非参法(PeptideProphet)还是机器学习法(Percolator)都依赖于较大数据集的统计评估,因此小规模数据集不适用该方法来提高灵敏度。
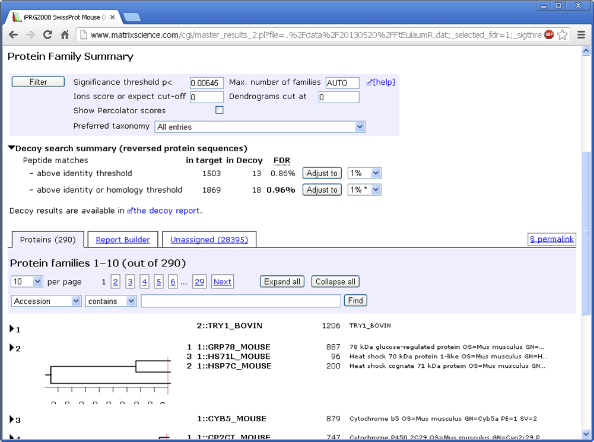
选择Decoy后,Mascot 会自动进行肽段FDR评估,此参数很多杂志审稿时都要求报告。通过将序列数据库中的序列反转或者随机重排来评估搜索结果的FDR阈值。在查看结果时,您可以调整预期的FDR为5%或1%,0.1%,1%,5%的FDR值是常用的评估大数据集的指标。注意,Mascot中调整FDR是评估肽段FDR,而非蛋白的FDR。
14、Report
Report选项会限制结果匹配的最大数量。目前我们大部分情况下选择AUTO即可。
15、参数设置小结
1、搜索参数非常关键,必须以精确的标准设定。
2、当搜索单个物种的数据库时,通常需要包含一个污染物数据库。
3、目标样品组成简单时,也要尽量选取来源物种全库搜索
4、只能指定高丰度的修饰为可变修饰。
5、如果蛋白被该蛋白酶消化,必须选择该蛋白酶。
6、使用error tolerant search 发现翻译后修饰,SNP,以及非特异性剪切产物。
7、对于重要的工作,运行target-decoy search, 将肽段的FDR 设为 1%, 筛选蛋白时,将匹配值设为2个不同的序列。

